
- 首页
- >
- GENESEED
- >
一个样本,三个世界:转录组vs翻译组vs蛋白组,你知道它们究竟有多不一样吗?
在现代生命科学研究里,转录组、翻译组和蛋白组已经成为解析细胞功能与调控的核心工具。它们分别记录了:基因被转录成mRNA的情况(转录组);哪些mRNA真正被核糖体翻译(翻译组);最终在细胞里“干活”的蛋白质(蛋白组)[1]。
听上去像一条顺畅的信息流:DNA→mRNA→正在被翻译的mRNA→蛋白。但现实是——同一个样本,三个层次讲的根本不是同一个故事。
要想真正看清细胞发生了什么,就必须跨越这三个世界,做协同分析;只有这样,才能更准确地捕捉到细胞内的生物学动态,为疾病早期诊断、治疗靶点发现,以及精准医学提供更扎实的数据支持[8–11]。
接下来,我们就从一个实际场景出发,用通俗易懂的语言,带你看懂这三个“世界”。
同一个样本,三个“故事”
试想这样一个场景:同一个样本,你做了三件“大事”——用RNA-seq做了转录组;用Ribo-seq/RNC-seq/polysome-profiling做了翻译组;用质谱做了蛋白组。当你把三份数据叠在一起时,会发现一个有点扎心的事实:转录组、翻译组和蛋白组,讲的根本不是同一个故事。
大量研究都在重复一个结论:mRNA水平和蛋白质水平通常只有中等相关,相关系数很多时候只有0.4–0.6,而且对单个基因来说,mRNA表达量高不代表蛋白丰度就一定高,两者差异可以相差好几个数量级[2–4,12]。也就是说——只看转录组去“脑补”蛋白变化,很容易翻车[2–4]。
那这三个“世界”到底差在哪?
翻译组跟蛋白组真的更接近吗?
我们先把三位主角介绍清楚,再看它们怎么协同。
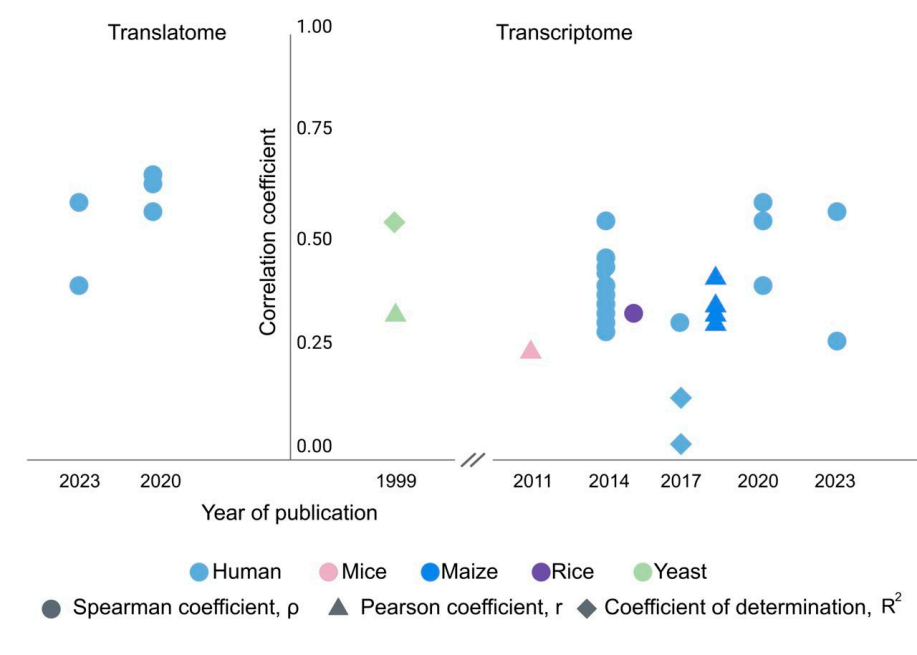
图1|基于转录组和翻译组数据的mRNA与蛋白质相关系数时间线[3]
三位主角分别管什么?
转录组:谁在“申请说话”?(潜力层)
转录组就是细胞里所有mRNA的全景图。它回答的是一个很基础的问题:“在当前条件下,哪些基因被转录了?表达量大概多少?”通常通过RNA-seq或测序芯片来实现。因为技术成熟、价格友好、分析体系完善,现在大量课题、项目只做到这一层就停了[1]。但转录组更多代表的是“表达潜力”,离真正的功能执行还隔着几道关卡。
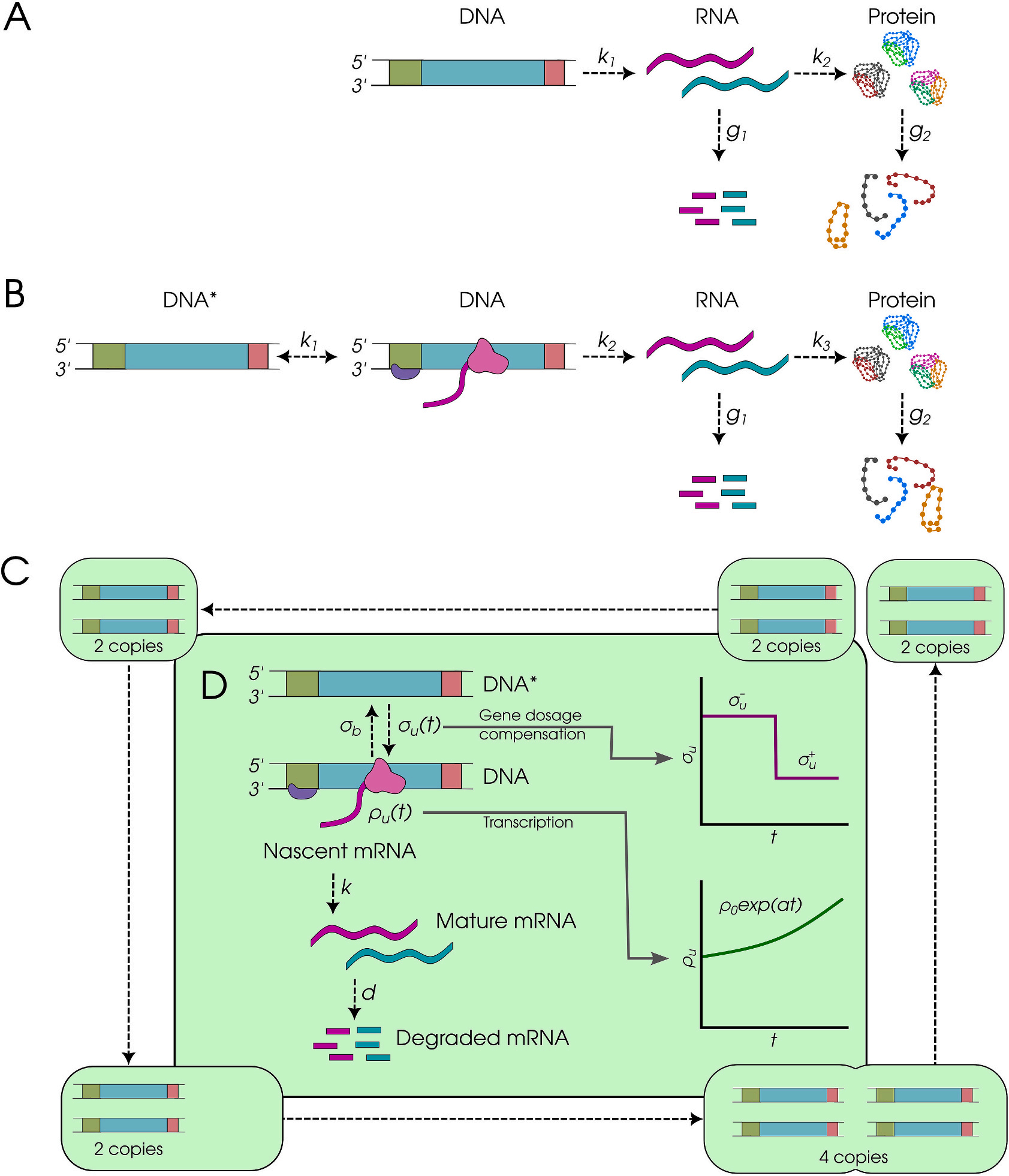
图2|随机基因表达模型[4]
太阳成集团tyc33455cc转录组服务
如果你正在做差异表达、发育轨迹、亚群划分等课题:从实验设计、样本建库到上机测序、数据分析,覆盖mRNA-seq、circRNA、全转录组等多种方案,提供差异分析、功能富集、通路分析等一整套报告,帮你把“谁在说话、说了多少”这件事交代清楚。

翻译组:谁真的“上台演讲”?(执行优先级)
翻译组关注的是:“在所有mRNA里,哪些是真正抢到核糖体资源,被优先翻译的?”典型手段包括:Ribo-seq(ribosome profiling):测核糖体保护片段,看到“被核糖体保护”的那段mRNA[6];RNC-seq(核糖体-新生肽链复合物测序)、polysome-profiling(多聚核糖体图谱分析)等。
这些技术能告诉你:哪些基因的翻译效率特别高或特别低;有没有“明明转录不高,却被重点翻译”的隐形关键基因[6];过去被认为是“非编码”的某些转录本,其实在产生短肽、小蛋白[6,7]。当你觉得“mRNA看着变了,但蛋白好像不太动”,翻译组就是那个能把故事讲完整的中间层[2,3]。
太阳成集团tyc33455cc翻译组(Ribo-seq/RNC-seq/polysome-profiling)平台
适合研究应激反应、肿瘤耐药、神经系统、免疫激活等强翻译调控场景[6–7];
能计算翻译效率(TE)、构建转录vs翻译对照矩阵;帮你挖掘隐藏ORF、短肽以及“翻译优先级”异常的基因[6,7]。

蛋白组:谁在“真正干活”?(功能终端)
蛋白质是细胞中的“工人”和“工具”,几乎所有功能执行——信号传导、代谢、结构支撑、应激响应——都要靠蛋白来完成[1]。蛋白组研究的就是:“在这个样本里,真正存在多少蛋白?都是什么?各有多少?”主要依赖质谱(LC-MS/MS)等技术进行定性和定量。
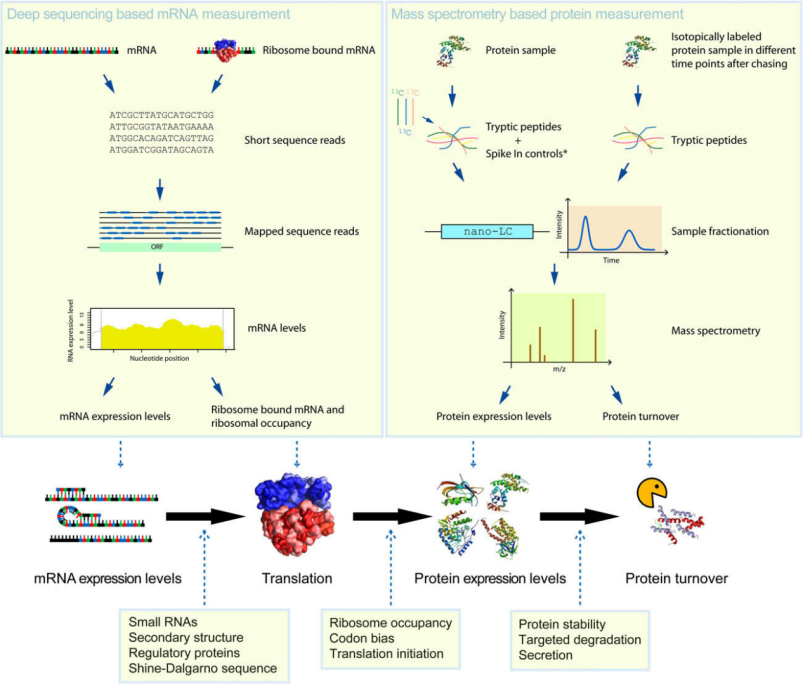
图3|分子生物学的中心途径[5]
太阳成集团tyc33455cc蛋白组学分析
提供Astral、4D DIA、TMT、label-free等多种定量策略;覆盖全蛋白组、靶向蛋白、磷酸化、泛素化等翻译后修饰;支持通路重构、网络分析、生物标志物筛选、潜在靶点评估[8,9]。
如果你的问题是:“到底哪些蛋白在变化?哪些节点是真的被药物/疾病打到了?”那答案可能在蛋白组,蛋白组学能直接回答蛋白质层面的变化,是解析药物靶点与疾病机制的核心证据之一[2,8–11]。

相关吗?远没有你想得那么高
mRNA vs蛋白:只有“中等关系”
大规模多组学研究普遍发现:在全基因组范围里,mRNA表达量与蛋白丰度的相关系数常在0.4–0.6左右;对很多基因来说,“mRNA表达量高≠蛋白丰度高”,同一个基因在不同条件下的mRNA/蛋白比值可以差几个数量级[2–4]。
这说明:转录组反映的是“潜力”,蛋白组呈现的是“现实”[1–4,12]。所以如果你只做转录组,然后在讨论里“顺嘴带一下蛋白”,其实风险是很大的[2–4,12]。
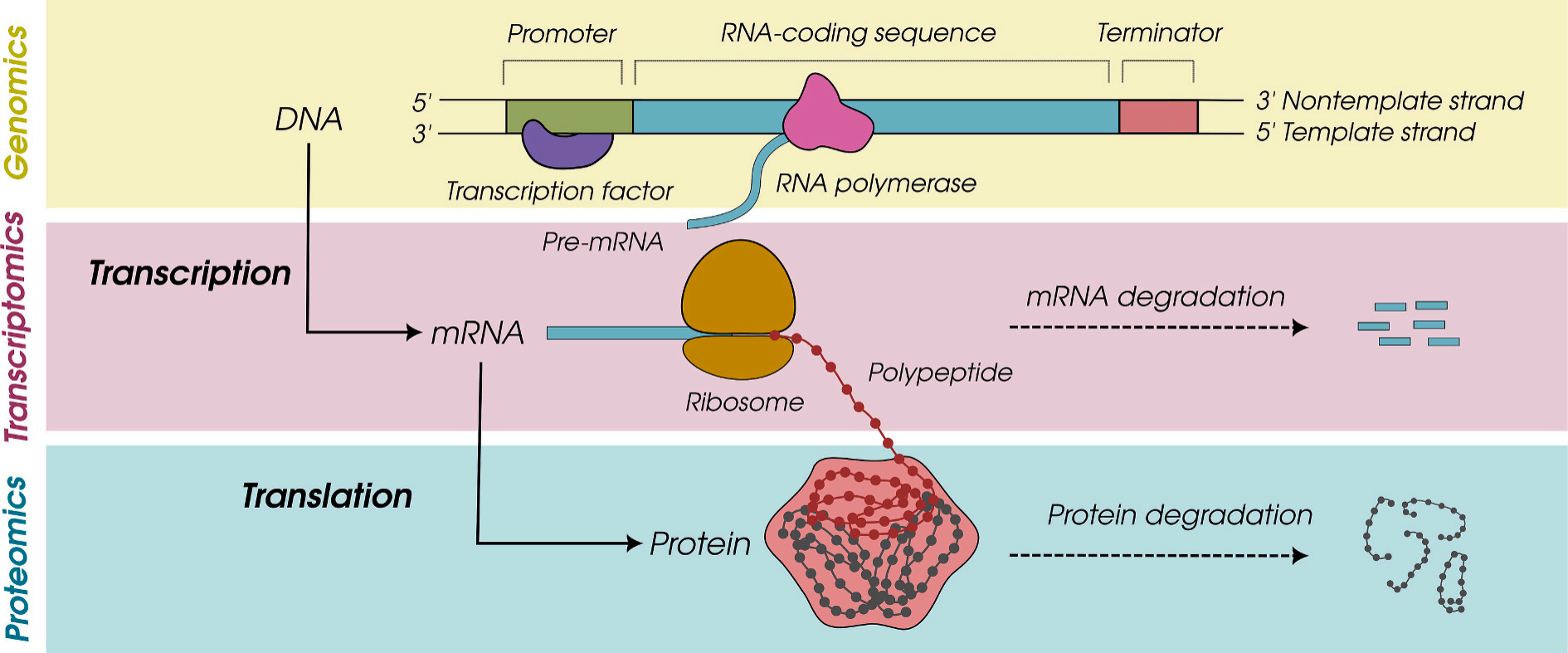
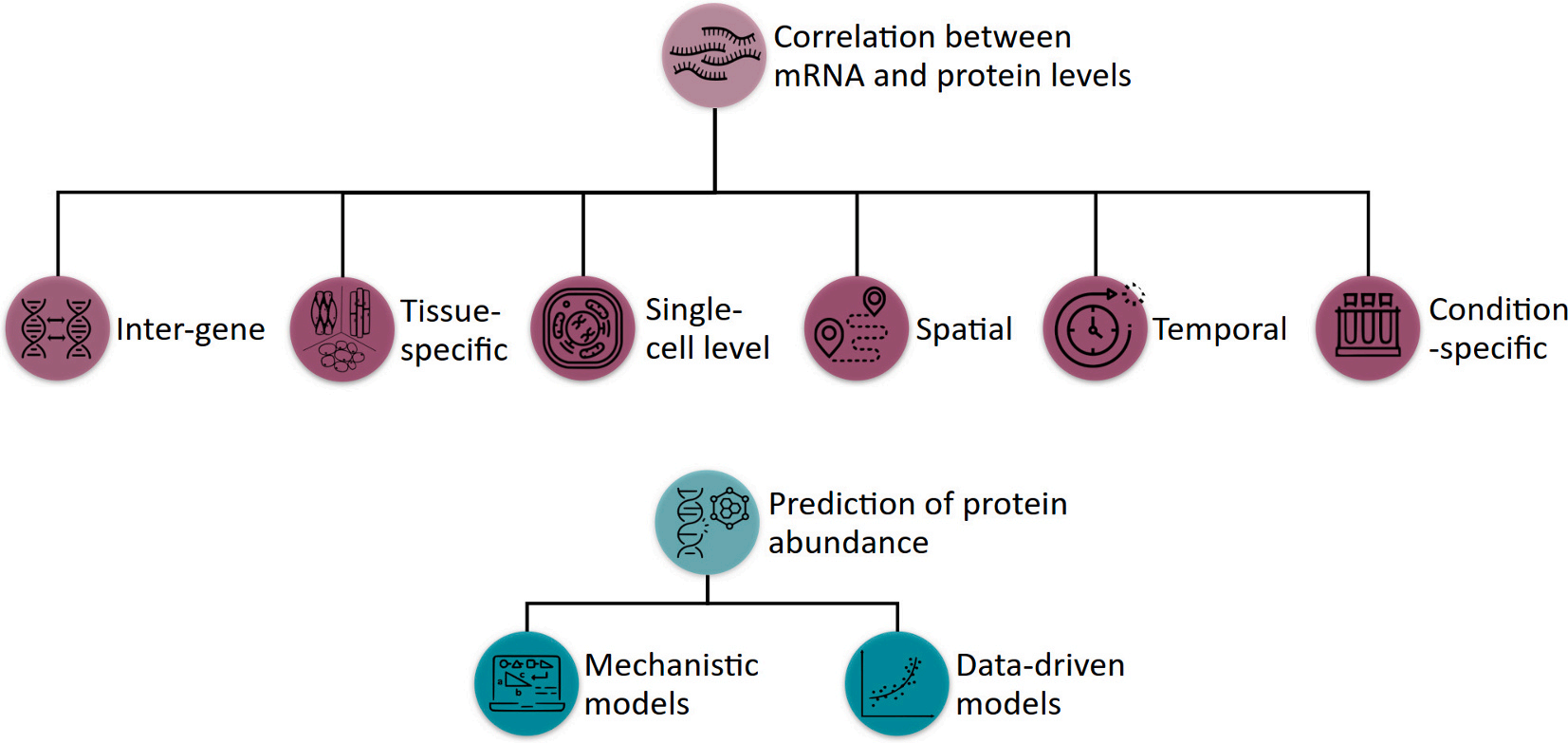
图4|分析蛋白质丰度和基因表达之间关系的研究分类标准[4]

图5|在大规模蛋白质组和转录组分析实验中观察到的mRNA表达量和蛋白质丰度之间的关系[1]
翻译组vs蛋白:确实近一点,但远非完美
很多人会问:“那加上翻译组,是不是就能比较准地预测蛋白了?”整体来说:单看mRNA,只能解释一部分蛋白丰度差异;把翻译效率(TE)+蛋白稳定性(半衰期)等信息一起纳入模型,对蛋白水平的解释度会明显提高[2–4];在不少系统中,翻译组与蛋白组的相关性确实高于转录组与蛋白组,但仍谈不上“可以直接互相替代”。
也就是说:转录组像“报名表”,翻译组像“到场名单”,蛋白组则是“最终在岗员工”[1–4,12]。
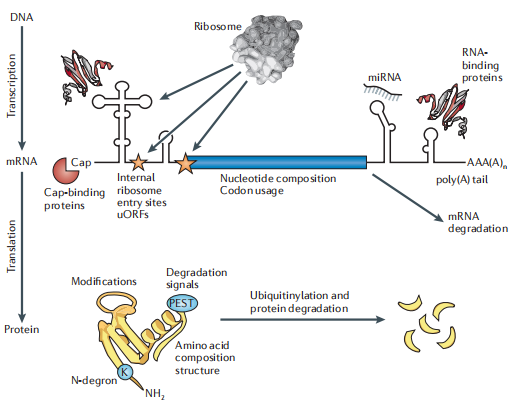
图6|翻译和蛋白质降解调控的模式[1]
一个样本三个结果:问题出在哪?
既然三层都是同一个样本,为什么结果差这么多?可以粗暴地理解为三道“过滤器”:
1
转录后调控:mRNA先被“精简一遍”
可变剪接产生不同isoform,有的基本不翻译;miRNA/lncRNA介导降解或翻译抑制;RNA结合蛋白影响mRNA稳定性、定位和可用性[1]。
这一步就足以让“测到的mRNA”和“真正参与翻译的mRNA”不再一一对应[1–3]。
2
翻译层调控:谁能抢到核糖体资源
翻译起始、延伸、终止都可能被精细调控:密码子偏好、tRNA供应、mRNA二级结构等都会影响翻译效率;应激、缺氧、药物处理下,细胞常常会大范围重排翻译优先级——一些“家务型基因”被压制,反而保留一小撮应激基因的翻译[6]。
如果你做的是药物处理、应激模型、免疫激活等课题,翻译组能帮你回答一个关键问题:“在资源有限的情况下,细胞究竟在重点翻译哪一批基因?”
3
蛋白层:稳定性、修饰和定位的再筛选
就算翻译出来了,蛋白依然可以被:快速降解(如泛素-蛋白酶体途径),做各类翻译后修饰(磷酸化、乙酰化、泛素化等),改变功能/稳定性;被运送到特定亚细胞位置,只在特定时空发挥作用[1,2]。
真正有价值的,是把这三个世界“串起来看”。
在疾病研究和精准医学里,多组学整合已经被证明有助于:更准确地分型和分层患者;更可靠地发现生物标志物;更精准地锁定干预靶点[8–11]。

图7|mRNA的表达量和蛋白丰度相关性研究的未来展望[4]
小结:看懂三个世界,用好一个样本
再回顾一次这三个层次的角色:
转录组:展示基因的表达潜力;
翻译组:告诉你哪些转录本真的被“动用”;
蛋白组:给出细胞实际的功能状态[1,3,13-14]。
三者共同构成了细胞内分子信息流动的完整链路,却绝不是简单的“一级传一级”。
要真正看清细胞发生了什么,必须跨越这三个世界做协同分析,这也是多组学在疾病研究和精准医学中越来越重要的原因[15]。
如果你正打算从“单组学”升级到“一个样本,三个世界”,欢迎来和我们聊聊你的研究方向、样本类型和预算,一起把这个样本里的三个世界,真正“看懂、用好、讲清楚”。

欢迎联系当地销售或拨打客服热线
参考资料









